Indice
Guida per l'utente - OBSOLETO vedi guida in Inglese
Accesso
Per poter accedere alle risorse occorre essere inseriti nel database LDAP del servizio HPC. Le richieste di accesso o in generale di assistenza vanno inviate a es_calcolo@unipr.it.
Una volta abilitati l'accesso avviene via SSH sull'host di login
ssh <nome.cognome>@login.hpc.unipr.it
L'accesso con password è consentito solo all'interno della rete di Ateneo (160.78.0.0/16) e della rete del Gruppo Collegato INFN di Parma (192.135.11.0/24). Al di fuori di questo contesto è necessario utilizzare la VPN di Ateneo oppure l'accesso con chiave pubblica.
Accesso password-less tra i nodi
Per utilizzare il cluster è necessario eliminare la necessità di utilizzare la password tra i nodi, utilizzando l'autenticazione a chiave pubblica. Occorre generare su login.hpc.unipr.it la coppia di chiavi, senza passphrase, e aggiungere la chiave pubblica nel file di autorizzazione (authorized_keys):
Generazione delle chiavi. Accettare i default premendo invio.
ssh-keygen -t rsa
Copia della chiave pubblica in authorized_keys:
cat ~/.ssh/id_rsa.pub >> ~/.ssh/authorized_keys
Accesso dall'esterno con chiave pubblica
Occorre generare la coppia di chiavi con il client SSH. La chiave privata dovrebbe essere protetta da una passphrase adeguata (non è obbligatorio ma è consigliato). La chiave pubblica deve essere inserita tra le vostre chiavi autorizzate (.ssh/authorized_keys) sull'host di login.
Se utilizzate il client SSH per Windows PuTTY (http://www.putty.org) è necessario generare la coppia di chiavi pubblica e privata con PuTTYgen e salvarle su un file. La chiave privata deve essere inserita nella configurazione di Putty (o WinSCP):
Configuration -> Connection -> SSH -> Auth -> Private key file for authentication
La chiave pubblica deve essere inserita nel file .ssh/authorized_keys di login.hpc.unipr.it
Link utili per la configurazione dei cliest SSH: Linux, MacOS X, PuTTY , Windows SSH Secure Shell
La chiave pubblica del client (esempio client_id_rsa.pub) deve essere inserita
nel file ~/.ssh/authorized_keys della macchina di login:
Copia della chiave pubblica in authorized_keys:
cat client_id_rsa.pub >> ~/.ssh/authorized_keys
Trasferimento di file
SSH è l'unico protocollo per la comunicazione con l'esterno e può essere utilizzato anche per il trasferimento dei file. In ambiente unix-like (Linux, MacOS X) si possono usare i comandi scp o sftp.
In ambiente Windows il programma più utilizzato è WinSCP (https://winscp.net/eng/docs/lang:it). In fase di installazione di WinSCP è possibile importare i profili di Putty.
SSH può essere utilizzato anche per il mount di un file-system remoto utilizzando SshFS (vedi http://www.fis.unipr.it/dokuwiki/doku.php?id=calcoloscientifico:guidautente#sshfs)
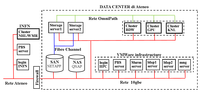
Hardware
L'attuale cluster è composto dai nuovi nodi di calcolo e dai nodi del preesistente cluster di Fisica / INFN.
Nuovi nodi di calcolo
- Cluster1 ( BDW)
- 8 nodi con 2 Intel Xeon E5-2683v4 (2x16 cores, 2.1GHz, 40MB smartcache), 128 GB RAM.
- 1 nodo con 2 Intel Xeon E5-2683v4 (2x16 cores, 2.1GHz, 40MB smartcache), 1024 GB RAM.
- Cluster2 ( GPU)
- 2 nodi con 2 Intel Xeon E5-2683v4 (2x16 cores, 2.1GHz), 128 GB RAM, 5 GPU NVIDIA P100-PCIE-12GB (Pascal architecture).
- Cluster3 ( KNL)
- 4 nodi con 1 Intel Xeon PHI 7250 (1x68 cores, 1.4GHz, 16GB MCDRAM), 192 GB RAM.
Dettaglio dei nodi:
Prestazioni di picco (doppia precisione):
1 Nodo BDW -> 2x16 (cores) x 2.1 (GHz) x 16 (AVX2) = 1 TFlops, Max memory Bandwidth = 76.8 GB/s 1 GPU P100 -> 4.7 TFlops 1 nodo KNL -> 68 (cores) x 1.4 (GHz) x 32 (AVX512) = 3 TFlops, Max memory bandwidth = 115.2 GB/s
Interconnessione con Intel OmniPath
Prestazioni di picco:
Bandwidth: 100 Gb/s, Latency: 100 ns.
Software
Il sistema operativo per tutte le tipologie di nodi è CentOS 7.X.
Ambiente Software (librerie, compilatori e tools): Elenco
Alcuni componenti software devono essere caricati per poter essere utilizzati.
Per elencare i moduli disponibili:
module avail
Per caricare / scaricare un modulo (esempio intel):
module load intel module unload intel
Per elencare i moduli caricati:
module list
Storage
Il nodo di login e i nodi di calcolo condividono le seguenti aree di storage:
| Mount Point | Env. Var. | Backup | Quota | Nota | Supporto |
|---|---|---|---|---|---|
| /hpc/home | $HOME | si | 50? GB | Programmi e dati | NAS |
| /hpc/group | $GROUP | si | 50? GB | Programmi e dati | NAS |
| /hpc/scratch | $SCRATCH | no | 1? TB, max 1 mese | Dati run-time | NAS high perf. |
| /hpc/archive | $ARCHIVE | no | Archivio | NAS / cloud | |
| /hpc/share | Software applicativo e database | NAS |
Acknowledgement
This research benefits from the High Performance Computing facility of the University of Parma, Italy ( HPC.unipr.it )
Gli autori sono pregati di comunicare i riferimenti delle pubblicazioni , che verranno elencati sul sito.
Sottomissione dei job con PBSpro
Le code sono gestite con PBSpro . I job vengono sottomessi sulla coda di routing e smistati automaticamente su una coda di esecuzione in base alle risorse richieste.
Comandi utili
Visualizza in forma sintetica lo stato delle code:
qstat -Q
Visualizza in dettaglio lo stato delle singole code:
qstat -Qf
Elenco dei nodi e loro stato:
pbsnodes -av
Per la sottomissione di un job:
qsub <opzioni> -I # modalità interattiva qsub <opzioni> script.sh # modalità batch
Visualizza i job in coda:
qstat
Opzioni principali
-q <nome coda>
Questa opzione seleziona la coda da utilizzare. Su nostro cluster non è necessaria poiché la coda di default (route) seleziona automaticamente la coda di esecuzione in base alle risorse selezionate.
-l <selezione risorse>
È l'opzione più importante poiché permette di indicare le risorse richieste:
- select: numero di chunk (gruppi di cores sullo stesso nodo)
- ncpus: numero di core per nodo (default 1)
- ngpus: numero di GPU per nodo
- mem: memoria richiesta per nodo
- mpiprocs: numero di processi MPI per nodo
- ompthreads: numero di thread OpenMP per processo
- place=scatter: possibilmente alloca i chunk su host separati (default)
- place=pack: possibilmente raggruppa i chunk sugli host
- place=excl: alloca gli host in modo esclusivo (non condivisi con altri job)
- cluster: seleziona il tipo di nodo (bdw | knl | gpu | fat | std)
Il cluster di default è std (standard) che sul cluster nuovo può essere bdw o fat, mentre sul cluster INFN può essere nhl o wmr.
Esempi di selezione delle risorse:
-l select=2:ncpus=2
-l walltime=<HH:MM:SS>
Tempo massimo di esecuzione del job. Questo dato seleziona la coda da utilizzare. Default: 24:00:00
Esempio:
-l walltime=00:30:00
-A <account name>
consente di specificare l'account su cui addebitare l'utilizza delle risorse. Esempi
-A nome.cognome -A G_HPC17A
-W group_list=<nome-gruppo>
consente di eseguire il job utilizzando il gruppo secondario specificato.
-j oe
redirige lo standard error sullo standard output.
-M <email>
L'opzione -M permette di indicare uno o più indirizzi e-mail, separati da virgola, che riceveranno le notifiche del gestore di code.
-m <opt>
L'opzione -m permette di indicare gli eventi che generano l'invio della notifica:
- a: notifica in caso di interruzione del job
- b: notifica all'avvio del job
- e: notifica alla fine del job
Esempio:
-M <nome.cognome>@unipr.it -m abe
Job interattivi
Per verificare l'elenco delle risorse assegnate si può utilizzare la sottomissione interattiva con opzione -I. Una volta entrati in modo interattivo il comando cat $PBS_NODEFILE visualizza l'elenco delle risorse assegnate. Il comando qstat -f lista maggiori dettagli riguardo le risorse assegnate.
qsub -I -l <regola di selezione> cat $PBS_NODEFILE qstat -f <job-id> exit
Esempi:
# 1 gruppo (chunk) di 2 CPU tipo STD qsub -I -l select=1:ncpus=2 # 2 chunk da 2 CPU di tipo KNL (possono stare sullo stesso nodo) qsub -I -l select=2:ncpus=2:cluster=knl # i chunk devono stare su nodi diversi qsub -I -l select=2:ncpus=2:cluster=knl,place=scatter # 1 chunk con 2 GPU sul cluster GPU qsub -I -l select=1:ngpus=2:cluster=gpu # 1 chunk da 1 cpu sul nodo wn51 e un chunk con 2 CPU sul nodo wn52 qsub -I -l select=1:ncpus=1:host=wn51:cluster=knl+1:ncpus=2:host=wn52:cluster=knl # 2 chunk ciascuno con 2 GPU su nodi diversi qsub -I -l select=2:ngpus=2:cluster=gpu,place=scatter:excl # mpiprocs dice a MPI quanti processi devono essere attivati per ogni chunk qsub -I -l select=2:ncpus=2:mpiprocs=1
Batch job
Occorre creare uno shell script che include le opzioni PBS e i comandi che devono essere eseguiti sui nodi.
per sottomettere il job:
qsub scriptname.sh
Ad ogni job viene assegnato un identificativo numerico univoco <Job Id>.
Al termine dell'esecuzione verranno creati i due file contenenti stdout e stderr nella directory da cui è stato sottomesso il job.
Per default i due file prendono il nome dello script con una estensione aggiuntiva:
Stdout: <script.sh>.o<job id> Stderr: <script.sh>.e<job id>
Job seriali, compilatore GNU
Compilazione dell'esempio mm.c per il calcolo del prodotto di due matrici:
cp /hpc/share/samples/serial/mm.* . g++ mm.cpp -o mm
Script mm.bash per la sottomissione dell'eseguibile seriale mm:
#!/bin/bash # Richiedi un chunk con 1 CPU #PBS -l select=1:ncpus=1 # Dichiara che il job durerà al massimo 30 minuti #PBS -l walltime=00:30:00 #stampa il nome del nodo assegnato cat $PBS_NODEFILE # Entra nella directory che contiene lo script cd "$PBS_O_WORKDIR" #esegui il programma ./mm
Sottomissione:
qsub mm.bash
Visualizza <job id> e lo stato:
qstat
Per cancellare eventualmente il job in esecuzione:
qdel <Job id>
Job seriali, compilatore Intel
Compilazione dell'esempio cpi_mc.c per il calcolo di Pi Greco:
cp /hpc/share/samples/serial/cpi/cpi_mc.c . module load intel icc cpi_mc.c -o cpi_mc_int
Script cpi_mc.bash per la sottomissione dell'eseguibile seriale cpi_mc_int:
#!/bin/bash #opzini PBS di default, si possono omettere #PBS -l select=1:ncpus=1 #PBS -l walltime=00:30:00 #stampa il nome del nodo assegnato cat $PBS_NODEFILE #carica il modulo del compilatore Intel module load intel # Entra nella directory che contiene lo script cd "$PBS_O_WORKDIR" #esegui il programma N=10000000 ./cpi_mc_int -n $N
Sottomissione:
qsub cpi_mc.bash
Job seriali, compilatore PGI
Compilazione dell'esempio cpi_sqrt.c per il calcolo di Pi Greco:
cp /hpc/share/samples/serial/cpi/cpi_sqrt.c . module load pgi pgcc cpi_sqrt.c -o cpi_sqrt_pgi
Script cpi_sqrt_pgi.bash per la sottomissione dell'eseguibile seriale cpi_sqrt_pgi:
#!/bin/bash # Opzioni PBS di default. Si possono omettere #PBS -l select=1:ncpus=1 #PBS -l walltime=00:30:00 cat $PBS_NODEFILE module load pgi cd "$PBS_O_WORKDIR" N=10000000 ./cpi_sqrt_pgi -n $N
qsub cpi_sqrt_pgi.bash
Job OpenMP con GNU 4.8
cp /hpc/share/samples/omp/omp_hello.c . gcc -fopenmp omp_hello.c -o omp_hello
Script omp_hello.bash con la richiesta di 12 CPU in uso esclusivo.
#!/bin/bash #PBS -l select=1:ncpus=32,place=excl #PBS -l walltime=00:30:00 #Unisci strerr con stdout #PBS -j oe cat $PBS_NODEFILE echo #OMP_NUM_THREADS : $OMP_NUM_THREADS cd "$PBS_O_WORKDIR" ./omp_hello
Job OpenMP con Intel
module load intel cp /hpc/share/samples/omp/mm/omp_mm.cpp .
Script mm_omp.bash con la richiesta di 1 nodo intero con almeno 32 core:
#!/bin/bash #PBS -l select=1:ncpus=32,place=excl #PBS -l walltime=00:30:00 #PBS -j oe #PBS -W group_list=<nome-gruppo> cat $PBS_NODEFILE cd "$PBS_O_WORKDIR" module load intel icpc -qopenmp omp_mm.cpp -o omp_mm # Per variare il numero di threads: export OMP_NUM_THREADS=8 echo OMP_NUM_THREADS : $OMP_NUM_THREADS ./omp_mm
Job OpenMP con PGI
cp /hpc/share/samples/omp/mm/omp_mm.cpp .
Script omp_mm_pgi.bash. Il cluster BDW è composto da nodi con 32 cores.
La variabile OMP_NUM_THREADS è per default pari al numero di core. Se vogliamo un numero di thread diverso lo possiamo indicare nella select:
#!/bin/sh #PBS -l select=1:ncpus=32:ompthreads=4:cluster=bdw #PBS -l walltime=00:30:00 #PBS -j oe cat $PBS_NODEFILE cd "$PBS_O_WORKDIR" module load pgi pgc++ -mp omp_mm.cpp -o omp_mm_pgi echo OMP_NUM_THREADS : $OMP_NUM_THREADS ./omp_mm_pgi
qsub omp_mm_pgi.bash
Job OpenMP con GNU 5.4
cp /hpc/share/samples/omp/cpi/* .
qsub cpi2_omp.bash
python cpi2_omp.py
Job MPI, GNU OpenMPI
module load gnu openmpi cp /hpc/share/samples/mpi/mpi_hello.c . mpicc mpi_hello.c -o mpi_hello
Script mpi_hello.sh per l'utilizzo della versione GNU OpenMPI:
#!/bin/bash # 4 chunk da 16 CPU ciascuno. Esegui un processo MPI per ogni CPU #PBS -l select=4:ncpus=16:mpiprocs=16 #PBS -l walltime=00:30:00 #PBS -j oe echo "### PBS_NODEFILE ###" cat $PBS_NODEFILE echo "####################" module load gnu openmpi cd "$PBS_O_WORKDIR" mpirun mpi_hello
qsub mpi_hello.bash
Job MPI con Intel MPI
module load intel intelmpi which mpicc cp /hpc/share/samples/mpi/mpi_mm.c . mpicc mpi_mm.c -o mpi_mm_int
Script mpi_mm_int.sh per l'utilizzo di Intel MPI:
#!/bin/sh # 4 chunk da 16 CPU ciascuno. Esegui un processo MPI per ogni CPU #PBS -l select=4:ncpus=16:mpiprocs=16 #PBS -l walltime=00:30:00 #PBS -j oe echo "### PBS_NODEFILE ###" cat $PBS_NODEFILE echo "####################" module load intel intelmpi cd "$PBS_O_WORKDIR" mpirun mpi_mm_int
Job MPI con PGI
module load pgi openmpi which mpicc cp /hpc/share/samples/mpi/mpi_hello.c . mpicc mpi_hello.c -o mpi_hello_pgi
Script mpi_hello_pgi.sh per l'utilizzo di openmpi di PGI:
#!/bin/sh #PBS -l select=4:ncpus=16 #PBS -l walltime=00:30:00 #PBS -j oe echo "### PBS_NODEFILE ###" cat $PBS_NODEFILE echo "####################" NPUSER=$(cat $PBS_NODEFILE | wc -l) module load cuda pgi openmpi cd "$PBS_O_WORKDIR" mpirun -hostfile $PBS_NODEFILE --npernode 1 mpi_hello_pgi
Job MPI + OpenMP con GNU OpenMPI
module load gnu openmpi cp -p /hpc/share/samples/mpi+omp/mpiomp_hello.c . mpicc -fopenmp mpiomp_hello.c -o mpiomp_hello_gnu
#!/bin/sh # 4 chunk da 16 CPU ciascuno, 1 processo MPI per ogni chunk, 16 thread OpenMP per processo MPI #PBS -l select=4:ncpus=16:mpiprocs=1:ompthreads=16 #PBS -l walltime=00:30:00 #PBS -j oe echo "### PBS_NODEFILE ###" cat $PBS_NODEFILE echo "####################" module load gnu openmpi cd "$PBS_O_WORKDIR" mpirun mpiomp_hello_gnu
Job MPI + OpenMP con Intel MPI
module load intel intelmpi cp /hpc/share/samples/mpi+omp/mpiomp-hello.c . mpicc -qopenmp mpiomp_hello.c -o mpiomp_hello_int
#!/bin/sh # 4 chunk da 16 CPU ciascuno, 1 processo MPI per ogni chunk, 16 thread OpenMP per processo MPI #PBS -l select=4:ncpus=16:mpiprocs=1:ompthreads=16 #PBS -l walltime=00:30:00 #PBS -j oe echo "### PBS_NODEFILE ###" cat $PBS_NODEFILE echo "####################" module load intel intelmpi cd "$PBS_O_WORKDIR" mpirun mpiomp_hello_int
Utilizzo del cluster KNL
Il compilatore da utilizzare è intel.
La selezione del cluster KNL avviene specificando cluster=knl come risorse richiesta.
Il massimo numero di core (ncpus) selezionabili per nodo è 68. Ogni core fisico include 4 core virtuali con tecnologia hyperthreading, per un totale di 272 per nodo.
#!/bin/sh # 4 nodi interi. Esegui un processo MPI per ogni nodo e 128 thread per processo #PBS -l select=4:ncpus=68:mpiprocs=1:ompthreads=128:cluster=knl #PBS -l walltime=00:30:00 #PBS -j oe echo "### PBS_NODEFILE ###" cat $PBS_NODEFILE echo "####################" module load intel intelmpi cd "$PBS_O_WORKDIR" cp /hpc/share/samples/mpi+omp/mpiomp_hello.c . mpicc -qopenmp mpiomp_hello.c -o mpiomp_hello_knl mpirun mpiomp_hello_knl
Utilizzo del cluster GPU
Il cluster GPU è costituito da 2 macchine con 5 GPU ciascuna. Le GPU di una singola macchina sono identificate da un ID numerico intero che va da 0 a 4.
Il compilatore da utilizzare è nvcc:
Esempio di compilazione:
cp /hpc/share/samples/cuda/hello_cuda.cu . module load cuda nvcc hello_cuda.cu -o hello_cuda
La selezione del cluster GPU avviene specificando cluster=gpu e ngpus=<1-5> tra le risorse richieste.
Esempio di sottomissione su 1 delle 5 GPU disponibili in un singolo nodo del cluster GPU:
#!/bin/sh # 1 nodo con 1 GPU #PBS -l select=1:ncpus=1:ngpus=1:cluster=gpu #PBS -l walltime=00:30:00 #PBS -j oe echo "### PBS_NODEFILE ###" cat $PBS_NODEFILE echo "####################" module load cuda cd "$PBS_O_WORKDIR" ./hello_cuda
Esempio di sottomissione del benchmark N-BODY su tutte e 5 le GPU disponibili in un singolo nodo del cluster GPU:
#!/bin/sh # 1 nodo con 5 GPU #PBS -l select=1:ncpus=1:ngpus=5:cluster=gpu #PBS -l walltime=00:30:00 #PBS -j oe echo "### PBS_NODEFILE ###" cat $PBS_NODEFILE echo "####################" module load cuda cd "$PBS_O_WORKDIR" /hpc/share/tools/cuda/samples/5_Simulations/nbody/nbody -benchmark -numbodies=1024000 -numdevices=5
Nel caso di N-BODY il numero di GPU da utilizzare viene specificato utilizzando l'opzione -numdevices (il valore specificato non deve superare quello del numero di GPU richieste con l'opzione ngpus).
In generale gli ID delle GPU da utilizzare vengono ricavati dal valore della variabile d'ambiente CUDA_VISIBLE_DEVICES.
Nel caso dell'ultimo esempio si ha
CUDA_VISIBLE_DEVICES=0,1,2,3,4
Utilizzo del cluster INFN
Attualmente il cluster INFN è gestito da un diverso Batch server: ce01.pr.infn.it
Per utilizzare il cluster INFN bisogna aggiungere:
#PBS -q route@ce01.pr.infn.it
In alternativa si può utilizzare l'host di login dell'INFN:
ssh login.pr.infn.it
Da login.pr.infn.it è possibile utilizzare il Batch server del nuovo cluster specificando:
#PBS -q route@ce01.hpc.unipr.it
Esempio mm:
cp /hpc/share/samples/serial/mm.cpp . g++ mm.cpp -o mm
Script di esecuzione mm.sh
#!/bin/sh #PBS -l select=1:ncpus=1 #PBS -l walltime=00:30:00 #PBS -j oe # Esegui il job utilizzando il seguente gruppo: #PBS -W group_list=T_RETI1617 # Esegui il job sul cluster INFN: #PBS -q route@ce01.pr.infn.it # Entra nella directory che contiene lo script cd "$PBS_O_WORKDIR" #stampa il nome del nodo assegnato cat " # $PBS_NODEFILE " ./mm -n 10000
Esecuzione:
qsub mm.sh
Verifica lo stato della coda:
qstat @ce01.pr.infn.it
Lavoro di gruppo
Per condividere i file tra i membri di un gruppo è necessario distinguere il tipo di attivita':
Nella modalità interattiva sul nodo di login il comando newgrp modifica il gruppo primario e i permessi dei nuovi file:
newgrp <nomegruppo>
Il comando newgrp sul cluster HPC esegue automaticamente anche il comando per entrare nella directory di gruppo ( /hpc/group/<nomegruppo> ) :
cd "$GROUP"
Nella modalità Batch occorre indicare il gruppo da utilizzare con la seguente direttiva:
#PBS -W group_list=<nomegruppo>
Scaling test
Per lanciare in sequenza una batteria di run in serie nello stesso job, ad esempio per verificare lo scaling di un algoritmo:
cp /hpc/share/samples/serial/cpi/cpi_mc.c . gcc cpi_mc.c o cpi_mc
Script launch_single.sh
#!/bin/bash cd "$PBS_O_WORKDIR" for N in $(seq 1000000 1000000 10000000) do CMD="./cpi_mc -n $N" echo "# $CMD" eval $CMD >> cpi_mc_scaling.dat done
qsub launch_single.sh
Gli output dei diversi run vengono scritti nel file cpi_mc_scaling.dat.
Per generare un plot dello scaling possiamo utilizzare la libreria matplotlib di python:
cp /hpc/share/samples/serial/cpi/cpi_mc_scaling.py .
python cpi_mc_scaling.py
Job Array
Utilizzando un'unico script PBS è possibile sottomettere una batteria di Job, che possono essere eseguiti in parallelo, specificando un parametro numerico diverso per ogni job sottomesso.
L'opzione -J consente di specificare la sequenza numerica dei parametri. Ad ogni lancio il valore del parametro è contenuto nella variabile $PBS_ARRAY_INDEX
Esempio:
Lancia N job per il calcolo di Pi greco con un numero di intervalli crescente da 100000 a 900000 con incremento di 10000:
cp /hpc/share/samples/serial/cpi/cpi_mc . gcc cpi_mc.c o cpi_mc
Script pbs_launch_parallel.sh
#PBS -J 100000-900000:10000
cd "$PBS_O_WORKDIR"
CMD="./cpi_mc -n ${PBS_ARRAY_INDEX}"
echo "# $CMD"
eval $CMD
qsub pbs_launch_parallel.sh
Riunisci gli output:
grep -vh '^#' pbs_launch_parallel.sh.o*.*
Job MATLAB
Esecuzione di un programma seriale MATLAB
cp /hpc/share/samples/matlab/pi_greco.m .
Script matlab.sh
#PBS -l walltime=00:30:00 #PBS -l select=1:ncpus=1 cd "$PBS_O_WORKDIR" module load matlab matlab -nodisplay -r pi_greco
qsub matlab.sh
Esecuzione di un job parallelo con MATLAB
cp /hpc/share/samples/matlab/pi_greco_parallel.m .
Script matlab_parallel.sh. La versione di Matlab installata sul cluster consente l'utilizzo massimo di 12 cores dello stesso nodo.
#PBS -l walltime=00:30:00 #PBS -l select=1:ncpus=12 cd "$PBS_O_WORKDIR" module load matlab matlab -nodisplay -r pi_greco_parallel
qsub matlab_parallel.sh
Esecuzione di un programma MATLAB su GPU
cp /hpc/share/samples/matlab/matlabGPU.m . # da fare
Script matlabGPU.sh
#PBS -l walltime=00:30:00 #PBS -l select=1:ncpus=1:ngpus=1 cd "$PBS_O_WORKDIR" module load matlab cuda matlab -nodisplay -r matlabGPU.m
qsub matlabGPU.sh
Job MPI Crystal14
Script crystal14.sh per la sottomissione della versione MPI di Crystal14. Richiede 4 nodi da 8 core ed avvia 8 processi MPI per nodo:
#!/bin/sh #PBS -N crystal14 #PBS -l select=4:ncpus=8:mpiprocs=8 #PBS -l walltime=168:00:00 # input files directory CRY14_INP_DIR='input' # output files directory CRY14_OUT_DIR='output' # input files prefix CRY14_INP_PREFIX='test' # input wave function file prefix CRY14_F9_PREFIX='test' source '/hpc/software/bin/hpc-pbs-crystal14'
Si consiglia di creare una cartella per ogni simulazione. In ogni cartella deve essere presente una copia dello script crystal14.sh.
- CRY14_INP_DIR: il file o i file di input devono trovarsi nella sottocartella 'input' della cartella corrente. Per utilizzare la directory corrente commentare la riga con la definizione della variabile CRY14_INP_DIR. Per cambiare sottocartella modificare il valore della variabile CRY14_INP_DIR.
- CRY14_OUT_DIR: i file di output verranno creati nella sottocartella 'output' della cartella corrente. Per utilizzare la directory corrente commentare la riga con la definizione della variabile CRY14_OUT_DIR. Per cambiare sottocartella modificare il valore della variabile CRY14_OUT_DIR.
- CRY14_INP_PREFIX: il file o i file di input hanno un prefisso che deve coincidere con il valore della variabile CRY14_INP_PREFIX. La stringa 'test' è puramente indicativa e non corrisponde ad un caso reale.
- CRY14_F9_PREFIX: il file di input, con estensione 'F9', è il risultato di una precedente elaborazione e deve coincidere con il valore della variabile CRY14_F9_PREFIX. La stringa 'test' è puramente indicativa e non corrisponde ad un caso reale.
Lo script crystal14.sh include, a sua volta, lo script di sistema /hpc/software/bin/hpc-pbs-crystal14. Quest'ultimo non può essere modificato dall'utente.
Sottomissione dello script di shell
Spostarsi nella cartella che contiene crystal14.sh e lanciare il comando seguente per sottomettere lo script al job scheduler:
qsub ./crystal14.sh
Analisi dei file prodotti da Crystal14 durante l'esecuzione del job
Durante l'esecuzione del job viene creata una cartella temporanea tmp che contiene i due file:
nodes.par machines.LINUX
Il file nodes.par contiene i nomi dei nodi che partecipano al calcolo parallelo.
Il file machines.LINUX contiene i nomi dei nodi che partecipano al calcolo parallelo con una molteplicità pari al numero di processi MPI avviati sul nodo.
Per individuare le cartelle temporanee prodotte da Crystal14 durante l'esecuzione del job, lanciare il comando seguente direttamente dal nodo di login:
eval ls -d1 /hpc/node/wn{$(seq -s, 81 95)}/$USER/crystal/* 2>/dev/null
Per verificare il contenuto dei file prodotti da Crystal14 durante l'esecuzione del job, l'utente può spostarsi in una delle cartelle evidenziate dal precedente comando.
Al termine dell'esecuzione del job, i due file machines.LINUX e nodes.par vengono cancellati. La cartella temporanea tmp viene cancellata solo se è vuota.
Non è dunque necessario accedere con SSH ai nodi che partecipano all'elaborazione per verificare il contenuto dei file prodotti da Crystal14.
Job Gromacs
Per definire la variabile d'ambiente GMXLIB aggiungere le righe seguenti al file $HOME/.bash_profile:
GMXLIB=$HOME/gromacs/top export GMXLIB
$HOME/gromacs/top è puramente indicativo. Modificatelo secondo le vostre preferenze.
Job Gromacs OpenMP
Script mdrun_omp.sh per richiedere in esclusiva un nodo con 32 core ed avviare 16 thread OpenMP:
#!/bin/sh #PBS -l select=1:ncpus=32:ompthreads=16,place=excl #PBS -l walltime=24:00:00 test "$PBS_ENVIRONMENT" = 'PBS_BATCH' || exit cd "$PBS_O_WORKDIR" module load gnu openmpi source '/hpc/share/applications/gromacs/5.1.4/mpi_bdw/bin/GMXRC' gmx mdrun -s topology.tpr -pin on
Job Gromacs MPI ed OpenMP
Script mdrun_mpi_omp.sh per richiedere in esclusiva un nodo con 32 core ed avviare 8 processi MPI (il numero di thread OpenMP verrà calcolato automanticamente):
#!/bin/sh #PBS -l select=2:ncpus=32:mpiprocs=8,place=excl #PBS -l walltime=24:00:00 test "$PBS_ENVIRONMENT" = 'PBS_BATCH' || exit cd "$PBS_O_WORKDIR" module load gnu openmpi source '/hpc/share/applications/gromacs/5.1.4/mpi_bdw/bin/GMXRC' NNODES=$(cat $PBS_NODEFILE | sort -u | wc -l) NPUSER=$(cat $PBS_NODEFILE | wc -l) OMP_NUM_THREADS=$((OMP_NUM_THREADS/(NPUSER/NNODES))) mpirun gmx mdrun -s topology.tpr -pin on
Job Abaqus
Job Abaqus MPI
Esempio di script abaqus.sh per eseguire Abacus su 1 nodo, 32 core, 0 GPU:
#!/bin/bash # Le opzioni PBS di default si possono omettere. # select : numero dei nodi di calcolo # ncpus : numero dei core per nodo (max 32) # cluster : tipologia del nodo di calcolo # walltime: tempo di esecuzione stimato, max 240 ore (meglio una stima di poco superiore a effettiva) #PBS -l select=1:ncpus=32:cluster=bdw #PBS -l walltime=00:30:00 cat $PBS_NODEFILE # Moduli necessari per l'esecuzione di Abacus module load gnu intel openmpi cd "$PBS_O_WORKDIR" abaqus j=testverita cpus=32 # j= nomefile.inp
Job Abaqus MPI con GPU
Esempio di script abaqus-gpu.sh per eseguire Abacus su 1 nodo, 6 core, 1 GPU:
#!/bin/bash # Le opzioni PBS di default si possono omettere. # select : numero dei nodi di calcolo # ncpus : numero dei core per nodo (max 6) # cluster : tipologia del nodo di calcolo # walltime: tempo di esecuzione stimato, max 240 ore (meglio una stima di poco superiore a effettiva) #PBS -l select=1:ncpus=6:ngpus=1,cluster=gpu #PBS -l walltime=00:30:00 cat $PBS_NODEFILE # Moduli necessari per l'esecuzione di Abacus module load gnu intel openmpi cuda cd "$PBS_O_WORKDIR" abaqus j=testverita cpus=6 gpus=1 # j= nomefile.inp
SSHFS
Per scambiare dati con una macchina remota su cui è installato un server ssh è possibile utilizzare SSHFS .
SSHFS è un file-system per sistemi operativi Unix-like (MacOsX, Linux, BDS). Questo file system permette di montare in locale una cartella posizionata su un host in cui gira SSH server. Questo software implementa il modulo del Kernel FUSE.
Attualmente è installato solo su login.pr.infn.it. In alternativa può essere installato sulla macchina linux remota per accedere ai propri dati sul cluster.
Per utilizzarlo:
mkdir remote # creiamo il mount-point sshfs <remote-user>@<remote-host>:<remote-dir> remote # mount del file-system remoto df -h # vedi i file-system montati ls remote/ fusermount -u remote # smonta il file-system
VTune
VTune e' un perfomance profiler di Intel ed e' disponibile sul cluster HPC.
Informazioni generali di Intel : https://software.intel.com/en-us/get-started-with-vtune-linux-os
Guida locale vtune (work in progress)